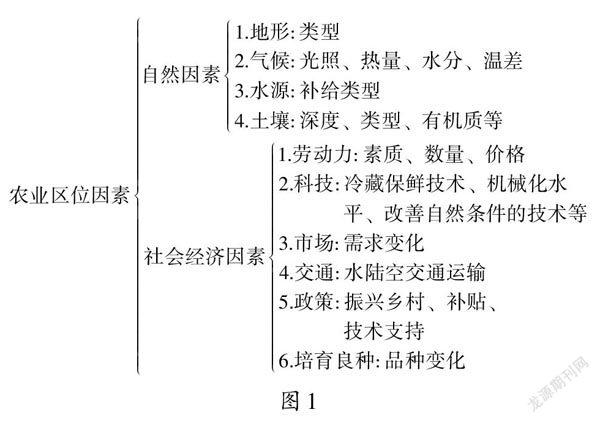
【www.zhangdahai.com--其他范文】
郭小蛟,唐玉明,2,田新惠,2,何劲松,刘成元,2,冯 军,2,刘茂柯,2,
(1.四川省农业科学院水稻高粱研究所,四川 德阳 618000;
2.四川省泸州市酿酒科学研究所,四川 泸州 646100)
中国白酒酿造依赖酒醅微生物的协同发酵。揭示酒醅微生物群落演替对白酒香味物质形成的影响是酿酒领域的研究热点。目前针对酒醅微生物的研究主要集中于宏基因组水平,宏蛋白质组学研究相对缓慢。蛋白质是基因功能的执行者,宏蛋白质组学是对微生物群落活动的直接表征。酒醅微生物宏蛋白质组学的匮乏阻碍了对白酒发酵机理的深入认识。
环境样品的理化性质复杂多样,无法形成统一的蛋白质提取方法。普遍情况下,环境样品需经液氮研磨和超声波破碎等方法使蛋白质充分释放后才能获得较好的提取效果。但不同于普通的环境样品,酒醅富含粮谷蛋白质,液氮研磨处理可能使其与微生物蛋白质混为一体。这可能导致在蛋白质的搜库鉴定中,将粮谷蛋白质误归于微生物,出现假阳性结果。在动物组织的宏蛋白质组学研究中,有报道称离心技术能有效分离动物组织和微生物菌体,降低宿主蛋白质对研究结果的影响。
基于此,本研究分别采用离心-超声波破碎法(centrifugation-ultrasonic disruption,C-UD)和液氮研磨-超声波破碎法(liquid nitrogen grinding-ultrasonic disruption,LNG-UD)提取酒醅微生物蛋白质,采用非标记定量蛋白质组学技术进行检测,比较两种样品预处理法获得的微生物群落结构差异,旨在为相关领域的深入研究提供一定依据。
1.1 材料与试剂
浓香型白酒酒醅样品采自泸州市某白酒企业。酿造结束后以窖池表面中心位置为采样点,利用课题组专利采集窖池上、中、下层酒醅,等量混合作为供试样品,-80 ℃保存。
甲酸、乙腈、质谱水、丙酮、蛋白标准品、苯甲基磺酰氟、SOLASPE 96 孔板、二喹啉甲酸(bicinchoninic acid,BCA)法蛋白质定量试剂盒美国Thermo Fisher Scientific公司;
乙二胺四乙酸二钠(ethylene diamine tetracetic acid disodium,EDTA-2Na)、甲醇、蔗糖、NaCl、二硫苏糖醇、聚丙烯酰胺、四乙基溴化铵、Tris-HCl 生工生物工程(上海)股份有限公司;
胰蛋白酶、碘代乙酰胺 美国Sigma-Aldrich公司;
DNA抽提试剂盒 美国MP Biomedicals公司;
Sensil 110 C-AQ色谱柱(250 mm×75 μm)澳大利亚IonOpticks公司。
1.2 仪器与设备
EASY-nLC1200液相色谱仪 美国Thermo Fisher Scientific公司;
Tims TOF Pro质谱仪 德国Bruker Daltonics公司;
十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)仪 美国Bio-Rad公司;
5810R台式冷冻离心机 德国Eppendorf公司。
1.3 方法
1.3.1 酒醅蛋白质提取
LNG-UD法:1 g酒醅样品经液氮研磨后加入离心管中,加入缓冲液(含0.70 mol/L蔗糖、0.01 mol/L NaCl、0.04 mol/L EDTA-2Na、0.01 mol/L二硫苏糖醇、0.13 mol/L pH 6.8 Tris-HCl、0.38 mol/L pH 8.8 Tris-HCl),加入苯甲基磺酰氟使其浓度为1 mmol/L。80 W冰上超声共3 min(超声1 s、间歇1 s)。
C-UD法:向1 g 不经液氮研磨的酒醅样品中加入缓冲液和苯甲基磺酰氟后超声波破碎(同LNG-UD法)。将上层漂浮的谷壳去除后,4 ℃、514×离心20 min,吸取中层溶液转移至新的离心管。
分别向两种预处理方法获得的溶液加入中等体积pH 7.8 Tris饱和酚,采用Tris/酚提取-甲醇/醋酸铵沉淀法提取蛋白质。每个处理设置3 个重复。
1.3.2 蛋白质定量和SDS-PAGE
采用BCA试剂盒进行蛋白质定量。从每个样品取10 μg蛋白质进行SDS-PAGE分析。
1.3.3 酶解及肽段除盐
将各处理3 次重复提取的蛋白质溶液等量混合,加入二硫苏糖醇使其终浓度为5 mmol/L,55 ℃孵育30 min。冷却后加入碘代乙酰胺使其终浓度为10 mmol/L,避光放置15 min。加入6 倍体积的丙酮于-20 ℃过夜。4 ℃、8 228×离心10 min,收集沉淀。加入 100 μL四乙基溴化铵复溶沉淀,以胰酶与蛋白质质量比1∶50加入胰酶,37 ℃过夜。酶解后采用SOLASPE 96 孔板脱盐。
1.3.4 液相色谱-质谱联用分析
将除盐后的肽段溶解于100 μL的流动相A,用EASY-nLC1200液相色谱仪进行分离。色谱条件:Sensil 110 C-AQ色谱柱(250 mm×75 μm);
流动相:A为0.1%甲酸溶液(/);
B为甲酸-乙腈-水溶液(0.1∶80∶19.9,/);
流速300 nL/min;
梯度洗脱:0~66 min,97%~73% A、3%~27% B;
66~73 min,73%~54% A、27%~46% B;
73~84 min,54%~0% A、46%~100% B;
84~90min,0% A、100% B。
质谱条件:毛细管电压1.4 kV;
干燥气温度180 ℃;
流速3 L/min;
扫描范围/100~1 700;
电喷雾离子源;
离子淌度范围0.6~1.6 cm/(V·s);
碰撞能20~59 eV。
1.3.5 蛋白质检索鉴定
质谱数据采用Proteome Discoverer 2.2在Uniprot数据库进行约束性和非约束性检索。检索参数:假阳性率0.01,漏切位点数2,固定修饰为脲甲基化;
可变修饰为氧化(M);
诱饵数据库模式为反向;
酶为胰蛋白酶;
质谱容忍度20;
特征肽段1。
1.3.6 扩增子测序
为指导蛋白质的约束性检索,对酒醅样品进行扩增子测序。采用总DNA抽提试剂盒提取酒醅微生物基因组。使用聚合酶链式反应(polymerase chain reaction,PCR)以引物5′-ACTCCTACGGGAGGCAGCA-3′和5′-CGGACTACHVGGGTWTCTAAT-3′扩增16S rRNA的V3/V4区;
以引物5′-GGAAGTAAAAGTCGTAACAAGG-3′和5′-GCTGCGTTCTTCATCGATGC-3′扩增基因。采用Illumina Novaseq平台对PCR产物进行双末端测序。PCR扩增、测序文库的构建及测序由上海派森诺公司完成。原始序列经Qiime 2软件优化处理,采用DADA 2将序列聚类为扩增子序列变异体后进行分类学注释,使用的数据库为SILVA_132(16S rRNA)和UNITE_8()。
1.4 数据处理

2.1 酒醅微生物蛋白质的SDS-PAGE分析
C-UD和LNG-UD预处理方法提取的酒醅蛋白质质量浓度差异不显著,分别为(4.19±1.21)g/L和(3.76±0.72)g/L。由图1可知,LNG-UD处理未见明显蛋白质条带,C-UD处理的蛋白质条带数量较多且清晰,表明两种预处理法提取的酒醅蛋白质质量有一定差异。

图1 酒醅微生物蛋白质SDS-PAGE图谱Fig.1 SDS-PAGE profile of microbial proteins from fermented grains for baijiu production
2.2 酒醅微生物蛋白质的鉴定信息
采用非约束和约束性两种检索方式在Uniprot数据库鉴定酒醅蛋白质。非约束性检索是指检索细菌和真菌的蛋白质库;
约束性检索是首先采用扩增子测序技术确定酒醅微生物的分类,再在纲水平检索11 个已知类群的蛋白质库(图2)。如图3所示,两种检索结果均显示两个预处理法获得的肽段和蛋白质种类差异显著,其中C-UD处理获得的酒醅肽段以及蛋白质数量均高于LNG-UD处理。如图4所示,C-UD处理在约束和非约束性检索中的蛋白质鉴定平均得分(Score Sequest HT)分别较LNG-UD处理提高了6.41%和15.11%。由表1可知,在两个处理检索分值排名前20位的蛋白质中,有13 个为两者共有;
其中除Q6BMK0和I1CB63,其余蛋白质在C-UD处理中的鉴定分值均高于LNG-UD。以上结果说明,C-UD处理蛋白质鉴定结果的可信度较高。


图2 基于扩增子测序的酒醅微生物群落结构Fig.2 Taxonomic classification of the microbial communities from fermented grains for baijiu production based on amplicon sequencing

图3 不同预处理方法的酒醅微生物肽段及蛋白质种类Venn图Fig.3 Venn diagram showing the number of unique and shared microbial peptides and proteins between extracts of fermented grains obtained by different pretreatment methods

图4 不同预处理方法的酒醅微生物蛋白质鉴定评分Fig.4 Score Sequest HT for proteins from fermented grains extracted by different pretreatment methods

表1 不同处理评分排名前20位的共有蛋白质Table 1 Top 20 shared proteins by Score Sequest HT
2.3 基于宏蛋白质组学的酒醅微生物群落结构
由图5a可知,酒醅中的细菌和真菌蛋白质数量基本一致,各约占蛋白质总量的50%。纲水平上,两种预处理方法在非约束性检索中获得的微生物群落结构差异显著。例如,C-UD处理的蛋白质主要属于Saccharomycetes和Bacilli,两者蛋白质数量占该组总量的59.79%,较LNG-UD处理显著提高,优势度更突出。此外,Schizosaccharomycetes是LNG-UD处理的第一优势纲,其蛋白质数量占该组总量的21.94%,但该类群在C-UD处理的优势度显著下降,蛋白质数量仅占该组总量的3.35%,排第7位。属水平上,两种预处理方法获得的优势菌属相似,在约束和非约束检索中均以和为最优势属;
不同点在于两者鉴定的前10位优势微生物的群落结构不同,也体现在菌属种类和数量的显著差异。由图5b可知,无论以何种方式检索,两个处理的共有菌属数量仅占各处理鉴定总量的61.83%~76.39%,而C-UD处理鉴定的菌属总量及其独有种类数量均高于LNG-UD处理。

图5 基于宏蛋白质组学的微生物群落结构(a)和属水平的Venn图(b)Fig.5 Microbial community structure (a) and Venn diagram showing the number of unique and shared microbial genera (b)based on metaproteomics
2.4 宏蛋白质组学和宏基因组学的酒醅微生物群落结构比较
由图2可知,扩增子测序技术从酒醅样品检出21 个已知细菌属和9 个已知真菌属,相对丰度大于1%的优势属为、和。由图6可知,两种预处理法在蛋白质水平上鉴定的菌属数量均高于扩增子测序。此外,两种技术检出的共有菌属仅占各自检测物种数量的7.50%~8.33%(宏蛋白质组学)和20%~33.33%(扩增子测序);
其中,C-UD处理和测序技术共有物种的数量高于LNG-UD,但两个处理均检出了测序技术检出的优势属和。

图6 宏蛋白质组学和宏基因组学鉴定微生物菌属的Venn图Fig.6 Venn diagram showing shared and unique microbial genera based on metaproteomics and metagenomics
3.1 不同预处理方法的酒醅微生物宏蛋白质组学差异分析
建立有效的蛋白质提取方法和检索适当的蛋白质数据库是宏蛋白质组学研究须解决的两个难点问题。研究认为,以基因组测序获得的物种分类为依据,选择相应物种的蛋白质数据库进行约束性检索可有效降低假阳性,提高结果的准确性。为全面比较两种预处理方法的提取效果,本实验采用约束性和非约束性检索两种方式。与以往窖泥宏蛋白质组学结果一致,非约束性检索鉴定的肽段和蛋白质数量均高于约束性检索(图3)。然而两种检索方式均显示,C-UD预处理法获得的酒醅肽段及蛋白质数量高于LNG-UD预处理法,且其蛋白质鉴定结果的可靠性较高(图3)。此外,在非约束检索中,两种预处理法获得的微生物群落结构显著不同(图5a)。这说明不同的预处理法对酒醅宏蛋白质组学的结果具有显著影响。这与以往肠道、厌氧发酵体系的宏蛋白质组学结果相似。Zhang Xu等比较不同蛋白质缓冲液(SDS、尿素、非离子洗涤剂)对人类肠道宏蛋白质组学的影响,发现用SDS缓冲液提取肠道蛋白质可显著提高蛋白质的产量以及肽段和蛋白质的鉴定数量;
该研究还显示,在不同缓冲液中加入钢珠进行敲打,均能使Firmicutes和Actinobacteria的相对丰度显著增加。该研究结果提示,除样品预处理方法外,酒醅蛋白质提取过程中的诸多环节还具有进一步优化的空间。
3.2 宏蛋白质组学和宏基因组学的酒醅微生物群落结构差异分析
实验结果显示,宏蛋白质组学和扩增子测序技术揭示的酒醅微生物群落结构具有显著差异。首先,两种技术检出的共有物种较少(图6)。其次在群落结构方面,扩增子测序技术检出的最优势真菌属未被宏蛋白质组学检出,而蛋白质水平上的最优势物种未被测序技术获得(图2、5)。此外,在测序中的丰度高达36.06%,但其蛋白数量在不同预处理方法的两种检索结果中均低于3 个,仅占蛋白质总量的0.33%~0.94%。除方法学因素外,造成以上差异的原因可能包括以下几点:1)某些微生物的蛋白质未被提取出或未被采用的技术检出;
2)部分微生物的蛋白质组未被充分揭示,Uniprot数据库尚缺乏相关蛋白质信息;
3)某些物种的基因丰度虽高但其生理功能可能被抑制,而一些低丰度物种也可能因生理活性较高而分泌大量蛋白质,行使重要功能。近年来,宏基因组测序技术被广泛用于酒醅微生物研究。其中,Qian Wei等和本实验的扩增子测序结果均显示,和是酒醅的最优势微生物。但在蛋白质水平上,本实验虽进一步证实了在酒醅中的优势地位,但未检出。以上基因组测序和宏蛋白质组学间的结果差异再次彰显出整合多组学研究的必要性;
为深入认识中国白酒的发酵机制,酿酒微生物的宏蛋白质组学研究亟待进一步加强。
分别采用C-UD法和LNG-UD法提取酒醅微生物蛋白质,采用非标记定量蛋白质组学技术进行检测,比较两种样品预处理法获得的微生物群落结构差异。结果表明,利用不同样品预处理法提取酒醅蛋白质对酒醅微生物宏蛋白质组学的结果具有重要影响。相对于LNG-UD法,C-UD法可提高酒醅肽段及蛋白质的鉴定数量,且其鉴定结果的可靠性较高;
C-UD处理鉴定的菌属总量及其独有种类数量均高于LNG-UD处理。后续研究将进一步优化酒醅微生物蛋白质的提取方法以获得最佳的提取条件,为酿酒微生物宏蛋白质组学的深入研究奠定一定基础。