【www.zhangdahai.com--其他范文】
方枫玲(综述),郭绍彬,傅亚,林曹瑞,刘灿,欧启水(审校)
感染性疾病严重威胁人类健康,也是临床工作的主要难题之一。分子诊断是感染性疾病诊断的重要部分。临床上常用的分子诊断方法为聚合酶链反应(polymerase chain reaction,PCR),但其对仪器、场地、人员的专业性要求限制了它在感染性疾病诊断中的应用。近年来,RNA调节系统被开发应用于分子诊断的研究日益增多[1-2]。立足开关(toehold switch)属于RNA核糖调控元件,它通过与目标RNA序列的碱基互补配对来控制基因表达[3]。2014年,哈佛大学GREEN团队[4]成功设计了立足开关,使其与无细胞转录翻译(cell-free transcription-translation,TX-TL)系统相结合,开创了分子诊断新的领域,成功实现了多种病毒性感染疾病相关病原体(如埃博拉病毒和寨卡病毒)的分子诊断[5-6]。
随着立足开关性能的提高,人们还将基于立足开关的体外无细胞转录翻译系统与纸基平台相结合,实现分子诊断的即时检验(point-of-care testing,POCT)[5-10]。随着立足开关技术的不断发展和完善,该技术必将给感染性疾病的现场快速诊断带来重大变革。
立足开关是一类基于RNA的从头设计的可编程原核生物核糖调控元件,其通过与任意序列的同源RNA相互作用进而诱导下游基因的表达。立足开关具有宽的动态范围、正交性和可编程性,通过联合无细胞转录翻译系统,可作为一种分子诊断方法应用于多种病原体的检测[4]。
1.1 立足开关的基本结构 传统的核糖调控系统通过自身RNA序列在核糖体结合位点(ribosome binding site,RBS)区域形成碱基互补配对来阻止核糖体结合,从而抑制基因的翻译。当与触发RNA(trigger RNA)结合时,RBS序列释放,启动基因的翻译。但是,由于抑制基因翻译是通过RBS序列结合实现的,因此触发RNA就必须含有RBS的序列,才能将抑制序列替换出来[4,11]。
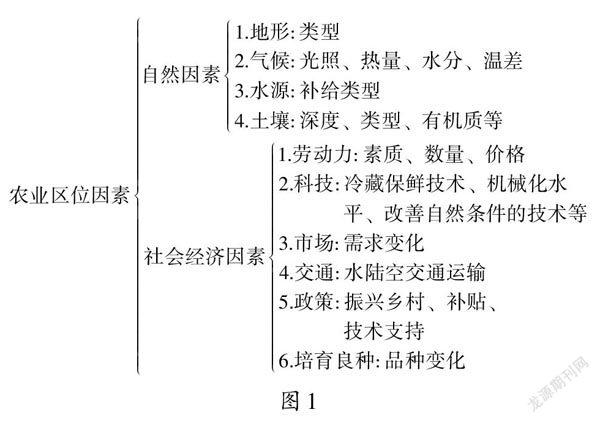
GREEN团队[4]提出一种解决方案,即设计立足开关,其可以结合和感知任意特定的RNA序列。立足开关是一个 RNA发夹结构。发夹结构5’末端的单链区域称为立足序列,可与触发RNA互补配对。开关包含受调控基因的编码序列,该编码序列的上游是基于发夹结构的信息处理模块,包含RBS和起始密码子,二者分别排列在一个环和一个凸起中,其后紧接一个具有21个核苷酸的连接序列(图1)。

RBS:核糖体结合位点;
AUG:起始密码子。图1 立足开关结构示意图Fig.1 Schematic view of the design of the toehold switch
与传统的核糖调控元件不同,立足开关通过在发夹环中隔离起始密码子(AUG)和RBS来阻断下游基因翻译,这个特性使立足序列不受限制,可设计任意的立足序列,用于不同病原体的分子诊断[4]。通过合理设计立足开关,使其结合任意特定的触发RNA序列,激活蛋白翻译,这不仅大大提高了序列的正交性,降低了串扰,而且触发RNA可靶向任意RNA序列的特性,显著拓展了立足开关的应用范围。
1.2 立足开关的设计原则 基于合成生物学的分子诊断系统通常由3个部分组成:传感器模块、信号处理器模块和报告模块[12]。对于立足开关而言,立足区域是传感器模块,中间的茎环结构是信号处理模块,下游的特定蛋白编码基因是报告模块。立足区域和报告基因可以根据所检测的病原体需求合理调整,并可以整合为更高复杂性的系统组件[5]。
设计立足开关应参照以下原则:(1)立足开关本身应形成一个稳定的发夹环结构,隔离RBS和起始密码子,避免在没有触发RNA存在的情况下激活下游基因表达而造成信号泄露;
(2)与单独存在的立足开关和触发RNA相比,立足开关-触发RNA复合体应有一个更稳定的能量状态,通过相互作用促进发夹环结构的展开和下游基因的激活;
(3)立足开关序列中不能包含禁止下游基因翻译的终止密码子;
(4)立足开关识别的触发RNA序列应是这个立足开关RNA特有的,以防止脱靶效应[13]。
1.3 立足开关的作用机制 当仅有立足开关存在时,其发夹结构将RBS与起始密码子AUG隔离,阻断下游编码基因翻译;
当触发RNA(病原体RNA)存在时,其与开关的立足区域结合,通过线性-线性杂交相互作用[14],启动 RNA-RNA链置换相互作用,打开发夹结构,释放RBS,从而激活基因并启始翻译[4]。通过将受调控的基因设计为报告基因,如GFP和lacZ基因,就可以可视化检测目标触发RNA分子的存在,这就是立足开关系统的分子诊断基础。
立足开关解决了传统核糖调控元件的设计局限性,具有宽动态范围、高正交性和高度可编程性。通过引入线性-线性杂交相互作用,而不是环-环或者环-线性作用,使得立足开关在动力学和热力学上比传统的核糖调控元件更有优势[4]。可以针对特定的病原体任意设计立足开关的立足序列,这种高灵活性有利于立足开关在分子诊断中的应用[15]。
1.4 无细胞转录翻译系统 无细胞转录翻译系统可以在体外控制平台上模拟体内细胞转录翻译的环境,从而在生物体外对生物系统进行原型设计和工程改造[16]。近年来,无细胞转录翻译系统为体外蛋白质表达以及 DNA调控元件、基因回路和代谢途径的原型设计提供了一个快速而有力的平台[17]。
无细胞转录翻译系统包含了体外基因表达所需的所有必要成分:(1)细胞提取物。主要通过细胞裂解并去除细胞碎片获得,包含核糖体、RNA聚合酶和其他从源菌株提取的转录翻译过程必需的蛋白质(如σ因子、起始因子和延伸因子)。(2)反应混合液。主要是蛋白合成的补充辅因子,包括氨基酸、核苷酸、盐、多胺和能量物质、拥挤试剂(如聚乙二醇或Ficoll)、代谢辅因子、缓冲液、转运 RNA(transfer RNA,tRNA)和其他代谢添加剂(例如谷胱甘肽)。(3)DNA模板。质粒或者线性表达模板(linear expression template,LET)。将3种成分混合并在16~37 ℃下孵育,体外DNA表达模板即可在细胞提取物和反应混合液的共同作用下,转录生成 mRNA,随后翻译成蛋白质,此过程通常需要1~24 h[18]。通过运用冷冻干燥技术,将配置好的无细胞转录翻译体系和基因模板分别固定在纸基或其他多孔材料上,在室温下可以长期储存长达1 a[9],易于运输携带。当二者混合并加水活化,基因模板便会在体系中表达,合成蛋白质[6]。
无细胞转录翻译系统的优势:(1)可大规模生产,成本低廉;
(2)可使用环状或易获取的PCR线性DNA模板;
可灵活利用各种辅助组分优化反应体系;
(3)体系可做成干粉状,易于储存和携带[6,19]。这些优势使无细胞转录翻译系统为核酸分子诊断平台提供一个强有力的技术支撑,大大提高了分子诊断技术的灵活性和便携性。目前,无细胞转录翻译系统已经应用于一些分子诊断领域,如寨卡病毒和呼吸道合胞病毒的分子诊断[1-2,5,10]。
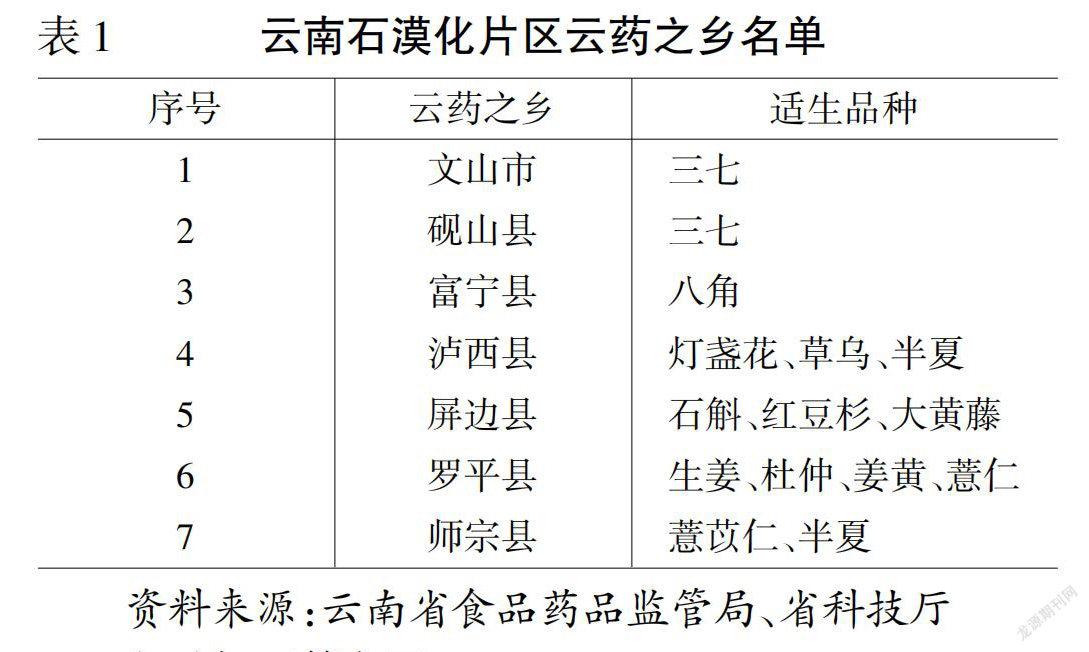
1.5 基于立足开关的无细胞转录翻译系统的工作原理 研究表明,立足开关常常在无细胞转录翻译系统中开发[5-6,8-9,20-21]。立足开关具有可以结合任意特定的RNA分子的能力,无细胞转录翻译系统具有在体外合成蛋白的能力,二者相结合,具有开发成分子诊断工具的潜力。将编码立足开关RNA的质粒DNA模板与无细胞转录翻译系统共同冷冻干燥至纸基上,加水活化后,质粒作为模板,在无细胞转录成分作用下,转录生成立足开关RNA。立足开关中的RBS被隔离,下游的报告基因处于抑制状态,在靶核酸存在的情况下,立足开关RNA与靶RNA互补配对,RBS被释放,激活下游的报告基因,在无细胞翻译成分的作用下,报告基因在体外翻译表达,通过联合不同的检测平台如荧光、比色和电化学等输出[4-6,22],在满足不同系统的要求下,便可以直观地检测报告基因的表达(图2)。根据以上工作原理,该系统已被开发应用于感染性疾病的分子诊断。

RBS:核糖体结合位点;
AUG:起始密码子。图2 立足开关工作原理图Fig.2 Schematic view of the operating principle of the toehold switch
根据基于立足开关的无细胞转录翻译系统作为分子诊断工具的工作原理,该系统已被开发成分子诊断技术,用于诊断多种感染性病原体,如埃博拉病毒、寨卡病毒和诺如病毒等[5-6,21],为感染性疾病的快速便携诊断提供了新的契机。
2014年,埃博拉疫情在西非爆发。PARDEE团队[6]开发了一种将无细胞基因合成网络嵌入到纸张的便携式低成本的诊断方法,通过将针对埃博拉病毒的立足开关传感器和无细胞转录翻译体系冷冻干燥在纸基上来快速检测埃博拉病毒。此开关仅需60~120 min 即可检测到埃博拉病毒RNA。另一方面,纸基传感器制造成本低廉,将其引入检测系统,使操作简单方便,可直接肉眼观察检测结果。
2015年,PARDEE团队[5]在无细胞纸基平台上开发了基于立足开关的RNA传感器,用于检测寨卡病毒。同时引入了一种可以与冻干传感器平台兼容的提供单碱基识别的检测方法,即NASBA-CRISPR裂解法(NASBA-CRISPR Cleavage,NASBACC),利用依赖核酸序列的扩增技术(nuclear acid sequence-based amplification,NASBA)的高效等温扩增和CRISPR/Cas9的序列特异性核酸酶活性来快速区分寨卡病毒的美洲谱系和非洲谱系。此外,该系统只需将病毒样品煮沸2 min,通过NASBA 扩增3 h,30 min便可以检测到猕猴血浆中病毒载量低至2.8×10-15mol/L的寨卡病毒。通过结合等温RNA扩增和冻干纸基平台,不仅提高了便携性,还降低了成本,该技术的检测试剂成本远低于目前临床核酸检测常用的实时荧光定量PCR技术(quantitative real-time PCR,qPCR),解决了基于核酸的分子诊断技术应用的局限性。以上这些优势使这项诊断技术以低廉的价格在资源匮乏地区的实施成为可能。
2018年,该团队在之前研究的基础上构建了新的方法,该方法可以在 3~5 h内特异性检测人类肠道微生物组中的10种细菌。通过结合NASBA,可以半定量检测艰难梭菌毒素的mRNA水平,因此可以快速区分艰难梭菌携带状态和艰难梭菌感染性腹泻[8]。同年,MA团队开发了另一种基于立足开关的无细胞转录翻译检测系统,可以从人类粪便标本中检测诺如病毒 GII.4型 Sydney流行株[21],在此基础上结合同源体的病毒富集和磁珠,将结合磁珠的检测限降低到2.7×10-19mol/L(灵敏度提高1 000倍)。2021年,PARK团队将逆转录环介导等温扩增法(reverse transcription loop-mediated isothermal amplification,RT-LAMP)结合到立足开关系统中,全程 70 min即可完成检测,检测限为120拷贝数/反应,达到了临床检测新型冠状病毒样本所需的灵敏度[7,23]。最近,国内的CAO和SUN团队基于立足开关的无细胞转录翻译系统,开发出多项可通过肉眼查看纸基显色、判断是否感染呼吸道合胞病毒以及区分其亚型的分子诊断技术[10,20]。
这些应用证明了基于立足开关的无细胞转录翻译系统在感染性疾病的分子诊断方面具有巨大潜力。相比临床上常用的qPCR技术,基于立足开关联合无细胞转录翻译系统的分子诊断平台,不仅表现出同样的特异度和灵敏度,而且不需要专门的检测仪器、专业的技术人员以及试剂的冷链运输条件,更加便携、快速、低成本[5-8,10,21],还能开发为试纸用于POCT,这些优势更加贴合感染性疾病诊断的要求。
目前,基于立足开关的无细胞转录翻译平台作为一种新兴的分子诊断技术,以便携、低成本、快速及高灵敏度等优势取得了较大进展,但该诊断技术也存在一些局限性,如立足开关的信号泄露,可能导致假阳性结果的产生;
检测平台无法实现完全定量。当前相关研究成果仅在实验室规模的环境中呈现,如果要将这种技术从实验室引入临床,并提供有效的诊断能力,还需要完善立足开关的设计、测试大规模样本,以及通过多学科整合对其效率和稳定性进行综合性评估。
目前,人们正在研究计算方法,以改进立足开关的设计方案和性能[24],通过多学科联合发展,不断地将新兴技术与该技术联合实现优势互补,以促进该诊断系统在临床实践中的应用。随着技术的发展,基于立足开关的无细胞转录翻译系统将使得针对临床常见感染性病原体,如生殖道感染病原体、呼吸道感染病原体等的快速检测成为可能,有利于我国POCT分子诊断的开发和应用,促进公共卫生事业的发展。
猜你喜欢 感染性病原体分子 《分子催化》征稿启事分子催化(2022年1期)2022-11-02去甲肾上腺素联合山莨菪碱治疗感染性休克的疗效观察中国典型病例大全(2022年12期)2022-05-13疫苗,原来是这么回事学生天地·小学中高年级(2018年10期)2018-12-13脂肪有时可助免疫恋爱婚姻家庭·养生版(2018年6期)2018-08-01“精日”分子到底是什么?新民周刊(2018年8期)2018-03-02米和米中的危险分子饮食科学(2017年12期)2018-01-02语文课堂教学的语言艺术考试周刊(2016年44期)2016-06-21拉肚子试试那些自然疗法母子健康(2015年5期)2015-11-20臭氧分子如是说少儿科学周刊·少年版(2015年1期)2015-07-07玻璃艺术中的病毒青少年科技博览(中学版)(2015年10期)2015-01-11本文来源:http://www.zhangdahai.com/shiyongfanwen/qitafanwen/2023/0428/590802.html